Dzisiaj przedstawiam dataset, do którego odnosić się będę w tematach klasyfikacji binarnej. Jest to zbiór danych zawierający skany histopatologiczne, które oznaczone są informacją o zawieraniu bądź niezawieraniu komórek rakowych. Zdjęć jest łącznie 327'680, co daje wiele możliwości w podejściu do dzielenia i wykorzystywania danych.
W tym poście krótko opiszę jak pobrać dane i co się tam znajduje.
[Ten post jest fragmentem serii "Krok po kroku" wprowadzającej do uczenia maszynowego (Machine Learning). Zapraszam do zapoznania się z całością.]
Pobieranie
Aby go ściągnąć odsyłam na stronę: https://github.com/basveeling/pcam
Do pobrania jest 9 plików:
- podzbiory: train/valid/test
- każdy w trzeb plikach: *x.h5.gz / *y.h5.gz / *meta.csv

Zawartość plików
W plikach z oznaczeniem "x" zawierają się skany (zdjęcia) o rozdzielczości 96x96 każde.
W plikach oznaczonych "y" zawierają się informacje o występowaniu komórek rakowych w centralnej części obrazu - dokładnie: środkowych 32x32 pikselach.
W plikach meta znajdują się dodatkowe informacje (dokładniej: poniżej), mniej istotnie z punku widzenia klasyfikacji.
Odczyt danych x,y
Aby odczytać dane obrazów i oznaczeń musimy zainstalować bibliotekę Python-a odczytującą format h5. Jest to h5py. W celu instalacji w linii komend należy wydać polecenie:
pip install h5pyOdczyt zawartości jest prosty, przykładowo:
import h5py
filename = "./camelyonpatch_level_2_split_valid_x.h5"
x = h5py.File(filename,'r+')['x']aby zobaczyć wymiar danych należy przeczytać wartość własności shape:
x.shapeWynik to: (32768, 96, 96, 3)
Czyli mamy do czynienia z 32768 zdjęciami (tutaj: zbioru walidacyjnego), każde o rozmiarze 96x96 i 3 kanałach kolorów.
Pojedyncze zdjęcie możemy wybrać indeksem, np:
img = x[0]Używając biblioteki PIL (Pillow) można takie przykładowe zdjęcie prosto zwizualizować:
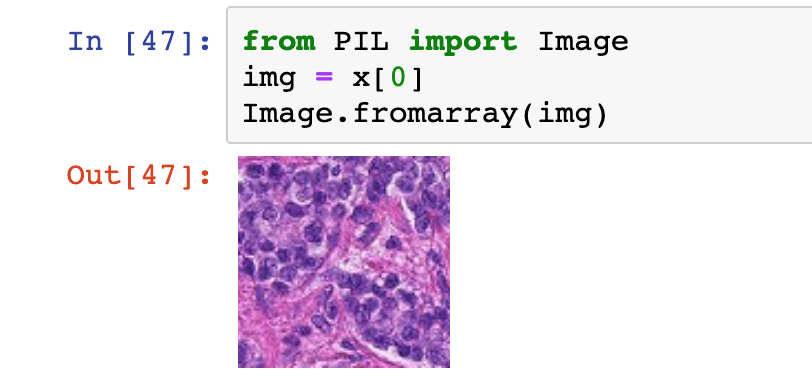
Opis występowania komórek rakowych w środkowych 32x32 pikselach określa nam oznaczenie na tożsamym indeksie w zmiennej y:
y[0]Wynik: array([[[1]]], dtype=uint8)
Wszystkie wyniki są równe 0 bądź 1.
Odczyt pliku meta
Plik meta najszybciej przeczytać za pomocą biblioteki Pandas:
import pandas as pd
meta = pd.read_csv("./camelyonpatch_level_2_split_valid_meta.csv")
meta.head()Po wpisaniu powyższych linii w Jupyter Notebook otrzymujemy podgląd pierwszych 5 wierszy:
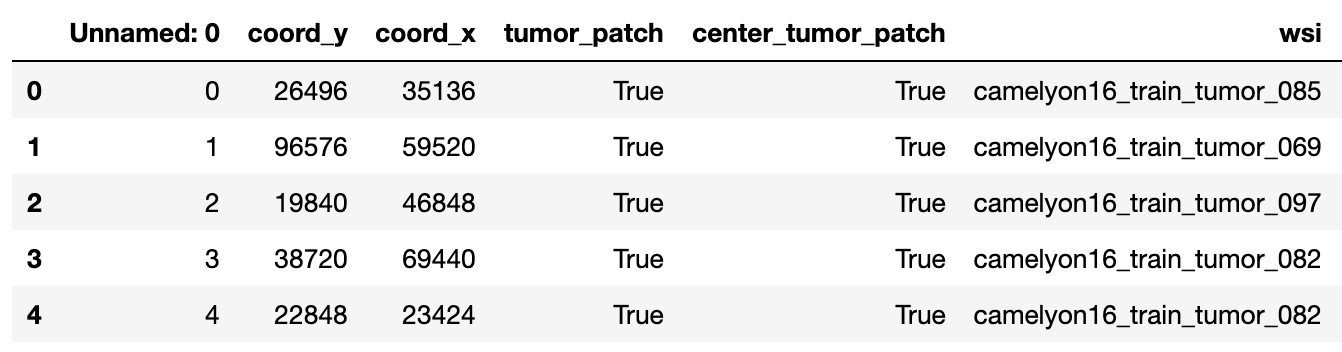
Dodatkową informacją jest występowanie komórek rakowych na zdjęciu, ale poza obszarem centralnym (tumor_patch=True oraz center_tumor_path=False).
Wartości y są równe center_tumor_patch.
Na stronie z danymi nie ma podanej jawnej interpretacji parametrów coord_y i coord_x, więc, mimo domysłów, na ten moment nie interpretujmy ich.
Co dalej?
Tego datasetu będę używać w kilku następnych postach to przedstawiania różnych konceptów Computer Vision.